
英语原文共 10 页
U-Net: 在细胞计数、检测和形态上的深度学习
Thorsten Falk1,2,3,19, Dominic Mai1,2,4.15.19, Robert Bensch1,2,16,19, Ouml;zguuml;n Ccedil;iccedil;ek1, Ahmed Abdulkadir1,5, Yassine Marrakchi1,2,3, Anton Bouml;hm1, Jan Deubner6,7, Zoe Jauml;ckel6,7, Katharina Seiwald6, Alexander Dovzhenko8,17, Olaf Tietz8,17, Cristina Dal Bosco8, Sean Walsh8,17, Deniz Saltukoglu2,4,9,10, Tuan Leng Tay7,11,12, Marco Prinz2,3,11, Klaus Palme2,8, Matias Simons2,4,9,13, Ilka Diester6,7,14, Thomas Brox1,2,3,7 and Olaf Ronneberger1,2,18*
对于经常发生的量化任务,例如在生物医学图像数据中的细胞检测和形状测量,U-Net是一种常用的深度学习解决方案。我们给出了一个ImageJ插件,它可以使非机器学习专家在无论是本地计算机还是远程服务器/云服务使用U-Net分析他们的数据。这个插件附带预训练模型,用于单细胞分割并允许U-Net在一些带注释样本的基础上适应新任务。
显微镜和样品制备技术的进步使研究人员获得了大量的图像数据。如果没有量化的障碍,这些数据可以提供更多的见解、更精确的分析、更严格的统计数据。在统计分析被应用之前,图像应该首先被转化为数字。通常,这种转化需要计数上千个带有确定标记的细胞或者画出细胞的轮廓去量化他们的形状或一个报告体的优势。这类工作很繁琐,因此经常被避免。例如,在神经科学研究中应用光遗传学工具,经常需要量化视蛋白表达神经元的数量或定位这些细胞中新开发的视蛋白。然而,因为需要付出努力,所以大多数发表的研究缺失这些信息。
这种量化不是计算机可以做的工作吗?确实如此。几十年来,计算机科学家已经开发出了专门的软件,可以解除生命科学研究人员的量化负担。但是,每个实验室都会生成不同的数据,并注重在当前研究问题的数据的不同方面。因此,必须为每种情况构建新软件。深度学习可以改变这种情况。由于深度学习从数据中学习任务的相关特征而不是被硬编码,因此软件工程师不需要为了某个具体量化任务设置专用软件。相反的,通用软件包能够学习去从适当的数据—生命科学研究人员能够自己提供的数据中自主地适应任务。
早在几年前,基于学习的方法引起了生物医学界的兴趣。常用的解决方案,例如ilastik1(http://ilastik.org/) 或可训练的WEKA分割工具包2(https://imagej.net/Trainable_Weka_Segmentation),可以通过使用通用手工定制图像特征训练细分管道。最近,注意力转移向深度学习,深度学习自动为实际图像分析任务提取最佳特征,从而避免了计算机科学专家对特征设计的需求3-5。然而,由于缺乏通用的、易于使用的软件包,生命科学中量化的广泛使用受到了阻碍。尽管Aivia(https://www.drvtechnologies.com/aivia6/) 和Cell Profiler6((http://cellprofiler.org/) 已经使用深度学习模型,但它们不允许对新数据进行训练,因此限制了它们在小部分数据集上的应用域。在图像恢复范围内,CSBDeep7提供了一个基于ImgLib2(https://imagej.net/ImgLib2)的插件,其中包含特定成像模式和生物标本的模型,并允许整合外部训练的新修复模型。另一种为生命科学带来深度学习的方法是CDeep3M8,它提供了一套命令行工具和教程,用于训练和应用残余初始-网络架构进行3D图像分割。这些软件包特别适合研究人员,通过提供不需要本地图形处理单元(GPU)的基于云端的设置,偶尔需要深度学习。
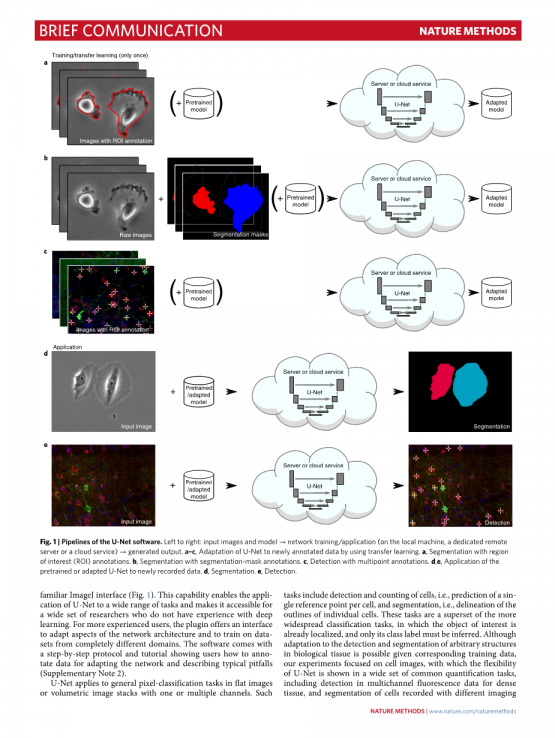
目前的工作提供了一种基于深度学习的通用软件包,用于细胞检测和细胞分割。对于我们成功的U-Net3编码器 - 解码器网络架构,它已经在生物医学数据分析基准测试9中取得了最高级别,并且已经成为生物医学图像分析中许多深度学习模型的基础,我们开发了一个作为插件运行在常用的ImageJ软件10中的接口(补充说明1和补充软件1-4)。与之前所有的软件包相比,我们的U-Net能够通过熟悉的ImageJ接口被用户自己训练并适应新的数据和任务(Fig.1)。这种功能使U-Net能够应用于广泛的任务,并使大量没有深度学习经验的研究人员可以使用它。对于更有经验的用户,该插件提供了一个接口,用于调整网络体系结构的各个方面,并在完全不同的域中训练数据集。该软件附带一步一步的协议和教程,向用户展示对于适应网络和描述典型陷阱如何注释数据(补充说明2)。
图1 | U-Net软件的管道。从左到右:输入图像和模型→网络训练/应用(在本地计算机上,专用远程服务器或云服务)→生成输出。a-c,通过使用转移学习使U-Net适应新注释的数据。a,利用感兴趣区域(ROI)注释进行分割。b,使用分割-掩模注释进行分割。c,使用多点注释进行检测。d,e,将预训练或适应的U-Net应用于新记录的数据。d,分割。e,检测。
U-Net适用于具有一个或多个通道的平面或立体图像堆栈中的一般像素分类任务。这样的任务包括检测和计数细胞,即预测每个细胞的单个参考点,以及分割,即描绘各个细胞的轮廓。这些任务是更广泛的分类任务的超集,其中感兴趣的对象已经被本地化,只是其类标签需要被推断出来。虽然在相应的训练数据下可以适应生物组织中任意结构的检测和分割,但我们的实验侧重于细胞图像,U-Net的灵活性在一系列常见的量化任务中得到展示,包括多通道检测密集组织的荧光数据,以及在2D和3D中用不同成像模式记录的细胞分割(图2和补充说明3)。在所有显示的案例中,U-Net将为科学家们节省大量工作。正如跨模式实验所示,生物样本的多样性对于单个软件来说太大而无法全部覆盖它们(补充说明3)。由于学习方法,U-Net的适用性从一组特殊情况扩展到无限数量的实验设置。对立体明亮区域数据进行极其精确的分割,其注释可以将人类专家推向极限,这是基于深度学习的自动量化软件功能的一个特别强有力的证明(图2d和补充说明3) 。
图2 | U-Net用于2D和3D检测和分割的示例应用。左边,原始数据;右边,U-Net输出(与2D情况下的人工注释比较)a,检测双通道落射荧光图像中的共定位。b,在五通道共聚焦图像堆栈中检测荧光蛋白标记的小胶质细胞。洋红色,所有小胶质细胞;红色,绿色和青色;五彩纸屑标记;蓝色,核染色。c,使用一个联合模式从荧光、差分干涉对比、相位对比和亮场显微镜等方面,在2D图像上进行细胞分割。d,3D亮场图像堆栈中的细胞分割。e,电子显微镜堆栈中的神经突分割。
批评者经常强调需要大量的培训数据来培训深层网络。在U-Net的情况下,基于不同数据集训练的基础网络和我们的特殊数据增强策略使得能够仅使用一个或两个带注释的图像来适应新任务(方法)。只有在特别困难的情况下才需要十多个训练图像。判断模型是否经过充分训练需要对稳定的验证集进行评估,并连续添加训练数据,直到无法观察到验证集的显著改进(补充说明3)。如果计算资源允许,随机分配的训练/测试拆分的交叉验证则避免了以多次训练为代价选择非代表性验证集。为使U-Net适应手头的任务而必须投入的人工工作通常远小于对实验数据进行最小统计分析所需的人工工作。此外,U-Net还可以在不需要任何额外工作的情况下运行更大的样品尺寸,从而使其特别适用于自动化大规模样品制备和记录设置,这些设备在未来几年可能会变得越来越普遍。
U-Net针对生命科学的可用性进行了优化。ImageJ中的软件集成和分步教程使得没有计算机科学背景的科学家可以深入学习(补充视频1-4)。重要的是,计算负荷对于普通生命科学实验室环境是可行的(补充说明1)。在具有消费者GPU的单台计算机上,网络适应新的细胞类型或成像模式的时间从几分钟到几小时不等。如果实验室中没有专用计算硬件,可以使用通用云服务。
我们的实验证明U-Net产生的结果与手动注释有类似的质量。与其他自动注释工具相比,U-Net的一个特征是注释的个体影响。该特征是有利的,因为研究人员通常开发单独的协议,其中在没有明确提及的情况下考虑多个参数。由于它们的复杂性,这些标记规则不能通过常用的自动标记工具再现。然而,这种优势也可能是一个缺点:U-Net从提供的例子中学习。如果示例不代表实际任务,或者这些示例中的手动注释质量低且不一致,则U-Net将无法训练或将重现新数据符合要求的注释。这方面也可以作为手动注释的质量检查。总的来说,U-Net无法校正低质量的人类注释,但却是将单个标注规则应用于大型数据集的工具,从而可以在各种量化任务中节省手动注释工作。
在线内容
任何方法,其他参考文献,自然研究报告摘要,源数据,数据可用性声明和相关的收藏代码均可在以下网站获取https://doi.org/10.1038/s41592-018-0261-2。
收到日: 2018年7月26日;采纳日: 2018年11月19日;
在线出版: 2018年12月17日
参考文献
1. Sommer, C, Strauml;hle, C, Koethe, U. amp; Hamprecht, F. A. in Ilastik: interactive learning and segmentation toolkit in IEEE Int. Symp. Biomed. Imaging. 230–233 (IEEE: Piscataway, NJ, USA, 2011).
2. Arganda-Carreras, I. et al. Bioinformatics 33, 2424–2426 (2017).
3. Ronneberger, O., Fischer, P. amp; Brox, T. U-Net: convolutional networks for biomedical image segmentation. in Medical Image Computing and Computer-Assisted Intervention—MICCAI 2015 Vol. 9351, 234–241 (Springer, Cham, Switzerland, 2015).
4. Rusk, N. Nat. Methods 13, 35 (2016).
5. Webb, S. Nature 554, 555–557 (2018).
6. Sadanandan, S. K., Ranefall, P., Le Guyader, S. amp; Wauml;hlby, C. Sci. Rep. 7, 7860 (2017).
7. Weigert, M. et al. Nat. Methods https://doi.org/10.1038/s41592-018-0216-7 (2018).
8. Haberl, M. G. et al. Nat. Methods 15, 677–680 (2018).
9. Ulman, V. et al. Nat. Methods 14, 1141–1152 (2017).
10. Schneider, C. A., Rasband, W. S. amp; Eliceiri, K. W. Nat. Methods 9, 671–675 (2012).
致谢
这项工作获得的支持有:在德国教育和研究部(BMBF)的下的MICROSYSTEMS项目(0316185B),与T.F. 和 A.D.相关;2012年伯恩斯坦奖(01GQ2301),与 I.D.相关;联邦经济事务和能源部(ZF4184101CR5),与A.B.相关; 德国研究基金会 (DFG) 下的合作研究中心的 KIDGEM (SFB 1140) 与 D.M., Ouml;.Ccedil;., T.F. 和 O.R.相关,以及(SFB 746, INST 39/839,840,841)与 K.P.相关; 卓越集群
资料编号:[5959]
以上是毕业论文外文翻译,课题毕业论文、任务书、文献综述、开题报告、程序设计、图纸设计等资料可联系客服协助查找。


